Alan hedefli mutajenez
Alan hedefli mutajenez, bir genin DNA dizisinde ve herhangi bir gen ürününde spesifik ve kasıtlı değişiklikler yapmak için kullanılan bir moleküler biyoloji yöntemidir. Ayrıca alana özgü mutajenez veya oligonükleotide yönelik mutajenez olarak da adlandırılan bu, DNA, RNA ve protein moleküllerinin yapısını ve biyolojik aktivitesini araştırmak ve protein mühendisliği için kullanılır.
Alan hedefli mutajenez, mutasyonları DNA dizilerine sokarak DNA kitaplıkları oluşturmak için en önemli laboratuvar tekniklerinden biridir. Bölgeye yönelik mutajenez elde etmek için çok sayıda yöntem vardır, ancak oligonükleotit sentezinin maliyetlerinin azalmasıyla, yapay gen sentezi artık bazen bölgeye yönelik mutajenez yerine bir alternatif olarak kullanılmaktadır. 2013 yılından bu yana, prokaryotik bir viral savunma sistemine dayalı CRISPR / Cas9 teknolojisinin geliştirilmesi, genomun düzenlenmesine de izin vermiştir ve mutagenez, nispeten kolaylıkla in vivo gerçekleştirilebilir.[1]
Tarih
Radyasyon veya kimyasal mutajenler kullanılarak mutajenezdeki erken girişimler, bölgeye özgü olmayan rastgele mutasyonlar oluşturdu.[2] Nükleotidlerin analogları ve diğer kimyasallar daha sonra lokalize nokta mutasyonları[3] oluşturmak için kullanıldı, bu tür kimyasalların örnekleri aminopurin,[4] nitrosoguanidin[5] ve bisülfittir.[6] Alan hedefli mutajenez, 1974 yılında Charles Weissmann'ın laboratuvarında GC'nin AT'ye geçişini indükleyen bir nükleotid analoğu N4-hidroksisitidin kullanılarak gerçekleştirildi.[7][8] Bununla birlikte, bu mutagenez yöntemleri, elde edebilecekleri mutasyon türüyle sınırlıdır ve daha sonraki alan hedefli mutajenez yöntemleri kadar spesifik değildir.
1971'de Clyde Hutchison ve Marshall Edgell, küçük faj parçaları ϕX174 ve kısıtlama nükleazları ile mutantlar üretmenin mümkün olduğunu gösterdi.[9][10] Hutchison daha sonra 1978'de iş arkadaşı Michael Smith ile DNA polimeraz ile bir primer uzatma yönteminde oligonükleotidler kullanarak bölgeye yönelik mutajenez için daha esnek bir yaklaşım üretti.[11] Bu sürecin geliştirilmesindeki rolü için Michael Smith, daha sonra Ekim 1993'te, polimeraz zincir reaksiyonunu icat eden Kary B. Mullis ile Nobel Kimya Ödülü'nü paylaştı.
Temel mekanizma
Temel prosedür, kısa bir DNA primerinin sentezini gerektirir. Bu sentetik primer, istenen mutasyonu içerir ve mutasyon sahası etrafındaki şablon DNA'ya tamamlayıcıdır, böylece ilgilenilen gendeki DNA ile hibridize olabilir. Mutasyon, tek bir baz değişikliği (bir nokta mutasyonu) çoklu baz değişikliği silme veya ekleme olabilir. Tek iplikli primer daha sonra genin geri kalanını kopyalayan bir DNA polimeraz kullanılarak genişletilir. Bu şekilde kopyalanan gen, mutasyona uğramış bölgeyi içerir ve daha sonra bir vektördeki bir konakçı hücreye sokulur ve klonlanır. Son olarak, istenen mutasyonu içerip içermediklerini kontrol etmek için mutantlar DNA dizilemesi ile seçilir.
Yaklaşımlar
Tek primerli uzatma kullanan orijinal yöntem, düşük mutant verimi nedeniyle verimsizdi. Ortaya çıkan bu karışım, hem orijinal mutasyona uğramamış şablonu hem de mutant ve mutant olmayan progenlerin karışık bir popülasyonunu üreten mutant ipliği içerir. Ayrıca, kullanılan şablon, mutant iplik metillenmemişken metillenmiştir ve daha az mutantla sonuçlanan metillenmiş şablon DNA'yı destekleyen yanlış eşleşme onarım sisteminin varlığı nedeniyle mutantlar karşı seçilebilir. O zamandan beri, mutagenezin etkinliğini iyileştirmek için birçok yaklaşım geliştirilmiştir.
Yeni teknikler genlere bölgeye özgü mutasyonu sokmanın daha basit ve daha kolay yollarına izin verdiğinden, çoğu 2000'li yılların başından beri laboratuvarlarda nadiren kullanılmasına rağmen, bölgeye yönelik mutagenezi etkilemek için çok sayıda yöntem mevcuttur.[12]
Kunkel'in yöntemi
1985'te Thomas Kunkel, mutantlar için seçme ihtiyacını azaltan bir teknik geliştirdi.[13] Mutasyona uğratılacak DNA parçası, M13mp18 / 19 gibi bir fajmid içine yerleştirilir ve daha sonra iki enzim dUTPaz (dut) ve urasil deglikosidaz (udg) eksikliği olan bir E. coli suşuna dönüştürülür. Her iki enzim de, bakteri kromozomunu dCTP'nin dUTP'ye spontan deaminasyonu yoluyla mutasyonlardan koruyan bir DNA onarım yolunun parçasıdır. DUTPase eksikliği, hücrede yüksek seviyede dUTP ile sonuçlanan dUTP'nin parçalanmasını önler. Urasil deglikosidaz eksikliği, urasilin yeni sentezlenen DNA'dan uzaklaştırılmasını engeller. Çift mutant E. coli faj DNA'sını kopyaladığından, enzimatik mekanizma dTTP yerine dUTP'yi yanlış birleştirerek bazı urasiller (ssUDNA) içeren tek iplikli DNA ile sonuçlanabilir. SsUDNA, ortama salınan bakteriyofajdan ekstrakte edilir ve daha sonra mutajenez için şablon olarak kullanılır. Primer uzantısı için istenen mutasyonu içeren bir oligonükleotid kullanılır. Oluşan heterodubleks DNA, dUTP içeren bir ebeveyn mutasyona uğramamış zincirden ve dTTP içeren mutasyona uğramış bir zincirden oluşur. DNA daha sonra vahşi tip dut ve udg genlerini taşıyan bir E. coli suşuna dönüştürülür. Burada urasil içeren ebeveyn DNA zinciri bozulur, böylece ortaya çıkan DNA'nın neredeyse tamamı mutasyona uğramış iplikten oluşur.
Kaset mutagenezi
Diğer yöntemlerden farklı olarak, kaset mutagenezinin DNA polimeraz kullanılarak primer uzatma içermesine gerek yoktur. Bu yöntemde, bir DNA parçası sentezlenir ve daha sonra bir plazmite yerleştirilir.[14] plazmitdeki bir bölgedeki bir kısıtlama enzimi tarafından bölünmeyi ve ardından ilgili gendeki mutasyonu içeren bir çift tamamlayıcı oligonükleotidin plazmite bağlanmasını içerir. Genellikle plazmit ve oligonükleotitte kesen sınırlama enzimleri, plazmitin yapışkan uçlarına ve birbirine bağlanmasına izin veren aynıdır. Bu yöntem,% 100'e yakın verimlilikte mutantlar üretebilir, ancak mutasyona uğratılacak siteyi çevreleyen uygun kısıtlama alanlarının mevcudiyeti ile sınırlıdır.
PCR sahasına yönelik mutajenez
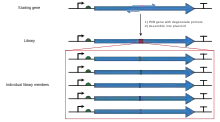
Kaset mutagenezinde kısıtlama alanlarının sınırlandırılması, oligonükleotid primerleri ile polimeraz zincir reaksiyonu kullanılarak aşılabilir, böylece iki uygun kısıtlama bölgesini kapsayan daha büyük bir parça üretilebilir. PCR'deki üssel amplifikasyon, daha sonra standart rekombinant moleküler biyoloji teknikleri kullanılarak orijinal içeriğe eklenebilen jel elektroforezi ile orijinal mutasyona uğramamış plazmitden ayrılacak yeterli miktarda istenen mutasyonu içeren bir fragman üretir. Aynı tekniğin birçok çeşidi vardır. En basit yöntem, mutasyon bölgesini parçanın uçlarından birine doğru yerleştirir, burada parçayı oluşturmak için kullanılan iki oligonükleotitten biri mutasyonu içerir. Bu, tek bir PCR adımını içerir, ancak yine de çok uzun bir primer kullanılmadıkça, mutasyon bölgesinin yakınında uygun bir kısıtlama alanı gerektirme gibi doğal bir soruna sahiptir. Bu nedenle diğer varyasyonlar, üç veya dört oligonükleotit kullanır; bunlardan ikisi, iki uygun kısıtlama bölgesini kapsayan ve sindirilebilen ve bir plazmite bağlanabilen bir fragman oluşturan mutajenik olmayan oligonükleotitler olabilirken, mutajenik oligonükleotit, içindeki bir lokasyona tamamlayıcı olabilir. herhangi bir uygun kısıtlama sitesinden çok uzakta parçalayın. Bu yöntemler, bağlanacak nihai parçanın istenen mutasyonu içerebilmesi için çok sayıda PCR adımını gerektirir. İstenen mutasyona ve ilgili kısıtlama alanlarına sahip bir fragman oluşturmak için tasarım süreci külfetli olabilir. SDM-Assist[15] gibi yazılım araçları süreci basitleştirebilir.
Tam plazmit mutagenezi
plazmit manipülasyonları için, diğer bölgeye yönelik mutagenez tekniklerinin yerini, büyük ölçüde, oldukça verimli, ancak nispeten basit kullanımı kolay ve bir kit olarak ticari olarak temin edilebilen teknikler almıştır. Bu tekniklerin bir örneği, bir çift tamamlayıcı mutajenik primerin, pfu polimeraz gibi yüksek kaliteli bir sarmal yer değiştirmeyen DNA polimeraz kullanılarak bir ısıl döngü reaksiyonunda tüm plazmiti büyütmek için kullanıldığı Quikchange yöntemidir.[16] Reaksiyon, çentikli, dairesel bir DNA oluşturur. Şablon DNA, metillenmiş DNA için spesifik olan DpnI gibi bir kısıtlama enzimi ile enzimatik sindirim yoluyla elimine edilmelidir. Escherichia coli suşlarının çoğundan üretilen tüm DNA'lar metillenecektir, E. coli içinde biyosentezlenen şablon plazmit bu nedenle sindirilecek ve in vitro olarak üretilen ve bu nedenle metillenmemiş olan mutasyona uğramış plazmit sindirilmemiş olarak bırakılacaktır. Bu çift sarmallı plazmit mutajenez yöntemlerinde, ısıl döngü reaksiyonu kullanılabilirken DNA'nın bir PCR'de olduğu gibi üssel olarak çoğaltılmasına gerek olmadığına dikkat edin. Bunun yerine amplifikasyon doğrusaldır ve bu nedenle zincir reaksiyonu olmadığından bunları bir PCR olarak tanımlamak yanlıştır.
Pfu polimerazın daha yüksek uzatma sıcaklığında (≥70 °C) sarmal yer değiştirebileceğini ve bu da deneyin başarısız olmasına neden olabilir, bu nedenle uzatma reaksiyonunun önerilen 68 °C sıcaklıkta gerçekleştirilmesi gerektiğini unutmayın. Bazı uygulamalarda, bu yöntemin çoklu primer kopyalarının eklenmesine yol açtığı gözlemlenmiştir. SPRINP adı verilen bu yöntemin bir varyasyonu, bu artefaktı önler ve bölgeye yönelik farklı mutagenez tiplerinde kullanılmıştır.[17]
Oligo yönelimli hedeflerin (SMOOT) tarama mutagenezi gibi diğer teknikler, plasmid mutagenezinde mutajenik oligonükleotitleri yarı rastgele birleştirebilir.[18] Bu teknik, tek mutasyonlardan bütün bir gen boyunca kapsamlı kodon mutagenezine kadar değişen plazmit mutagenez kitaplıkları oluşturabilir.
In vivo sahaya yönelik mutagenez yöntemleri
- Delitto perfetto[19]
- Yer değiştirme "pop-in pop-out"
- Doğrudan gen delesyonu ve bölgeye özgü mutagenez, PCR ve bir geri dönüştürülebilir işaretleyici ile
- Doğrudan gen delesyonu ve bölgeye özgü mutagenez, PCR ve uzun homolog bölgeler kullanılarak geri dönüştürülebilir bir işaretleyici ile
- Sentetik oligonükleotidlerle in vivo bölgeye yönelik mutagenez[20]
CRISPR
2013'ten beri CRISPR-Cas9 teknolojisinin geliştirilmesi, çeşitli mutasyonların çok çeşitli organizmaların genomuna verimli bir şekilde dahil edilmesine izin verdi. Yöntem, bir transpozon yerleştirme bölgesi gerektirmez, hiçbir işaret bırakmaz ve etkinliği ve basitliği, onu genom düzenleme için tercih edilen yöntem haline getirmiştir.[21][22]
Uygulamalar

Bölgeye yönelik mutajenez, geliştirilmiş veya özel özelliklere sahip rasyonel olarak tasarlanmış bir protein üretebilen mutasyonlar oluşturmak için kullanılır (yani protein mühendisliği).
Araştırma araçları - DNA'daki spesifik mutasyonlar, bir DNA sekansının veya bir proteinin fonksiyon ve özelliklerinin rasyonel bir yaklaşımla araştırılmasına izin verir. Ayrıca, proteinlerdeki bölgeye yönelik mutagenez ile tek amino asit değişiklikleri, çeviri sonrası değişikliklerin önemini anlamaya yardımcı olabilir. Örneğin, bir substrat proteininde belirli bir serinin (fosfoaseptör) bir alanine (fosfo-alıcı olmayan) değiştirilmesi, bir fosfat grubunun bağlanmasını bloke eder ve böylece fosforilasyonun araştırılmasına izin verir. Bu yaklaşım, CBP proteininin kinaz HIPK2 tarafından fosforilasyonunu ortaya çıkarmak için kullanılmıştır.[23] Diğer bir kapsamlı yaklaşım, bir kodonun veya bir dizi kodonun spesifik pozisyonlarda tüm olası amino asitlerle ikame edilebildiği alan doygunluk mutajenezidir.[24]
Ticari uygulamalar - Proteinler, belirli bir uygulama için uyarlanmış mutant formlar üretmek üzere tasarlanabilir. Örneğin yaygın olarak kullanılan çamaşır deterjanları, vahşi tip formu, işlemdeki proteinin aktivitesini önemli ölçüde azaltan ağartıcı ile oksitlenebilen bir metiyonine sahip subtilisin içerebilir.[25] Bu metiyonin, alanin veya diğer tortularla değiştirilebilir, bu da onu oksidasyona dirençli hale getirir ve böylece proteini ağartıcı varlığında aktif tutar.[26]
Gen sentezi
DNA oligonükleotid sentezinin maliyeti düştüğü için, tam bir genin yapay sentezi artık mutasyonu gen içine sokmak için uygun bir yöntemdir.
Bu yöntem, belirli bir organizma için onu optimize etmek için genin kodon kullanımının tamamen yeniden tasarlanması da dahil olmak üzere, çoklu alanlar üzerinde kapsamlı mutageneze izin verir.[27]
Kaynakça
- Hsu PD, Lander ES, Zhang F (Haziran 2014). "Development and applications of CRISPR-Cas9 for genome engineering". Cell. 157 (6): 1262-78. doi:10.1016/j.cell.2014.05.010. PMC 4343198 $2. PMID 24906146.
- Kilbey, B. J. (1995). "Charlotte Auerbach (1899-1994)". Genetics. 141 (1): 1-5. PMC 1206709 $2. PMID 8536959.
- Shortle, D.; Dimaio, D.; Nathans, D. (1981). "Directed Mutagenesis". Annual Review of Genetics. 15: 265-294. doi:10.1146/annurev.ge.15.120181.001405. PMID 6279018.
- Caras, I. W.; MacInnes, M. A.; Persing, D. H.; Coffino, P.; Martin Jr, D. W. (1982). "Mechanism of 2-aminopurine mutagenesis in mouse T-lymphosarcoma cells". Molecular and Cellular Biology. 2 (9): 1096-1103. doi:10.1128/MCB.2.9.1096. PMC 369902 $2. PMID 6983647.
- McHugh, G. L.; Miller, C. G. (1974). "Isolation and Characterization of Proline Peptidase Mutants of Salmonella typhimurium". Journal of Bacteriology. 120 (1): 364-371. PMC 245771 $2. PMID 4607625.
- D Shortle; D Nathans (1978). "Local mutagenesis: a method for generating viral mutants with base substitutions in preselected regions of the viral genome". Proceedings of the National Academy of Sciences. 75 (5): 2170-2174. doi:10.1073/pnas.75.5.2170. PMC 392513 $2. PMID 209457.
- R A Flavell; D L Sabo; E F Bandle; C Weissmann (1975). "Site-directed mutagenesis: effect of an extracistronic mutation on the in vitro propagation of bacteriophage Qbeta RNA". Proc Natl Acad Sci U S A. 72 (1): 367-371. doi:10.1073/pnas.72.1.367. PMC 432306 $2. PMID 47176.
- Willi Müller; Hans Weber; François Meyer; Charles Weissmann (1978). "Site-directed mutagenesis in DNA: Generation of point mutations in cloned β globin complementary DNA at the positions corresponding to amino acids 121 to 123". Journal of Molecular Biology. 124 (2): 343-358. doi:10.1016/0022-2836(78)90303-0. PMID 712841.
- Hutchison Ca, 3.; Edgell, M. H. (1971). "Genetic Assay for Small Fragments of Bacteriophage φX174 Deoxyribonucleic Acid". Journal of Virology. 8 (2): 181-189. PMC 356229 $2. PMID 4940243.
- Marshall H. Edgell, Clyde A. Hutchison, III, and Morton Sclair (1972). "Specific Endonuclease R Fragments of Bacteriophage X174 Deoxyribonucleic Acid". Journal of Virology. 9 (4): 574-582. PMC 356341 $2. PMID 4553678.
- Hutchison CA, Phillips S, Edgell MH, Gillam S, Jahnke P, Smith M (Eylül 1978). "Mutagenesis at a specific position in a DNA sequence" (PDF). J. Biol. Chem. 253 (18): 6551-60. PMID 681366.
- Braman, Jeff, (Ed.) (2002). In Vitro Mutagenesis Protocols. Methods in Molecular Biology. 182 (2nd bas.). Humana Press. ISBN 978-0896039100.
- Kunkel TA. (1985). "Rapid and efficient site-specific mutagenesis without phenotypic selection". Proceedings of the National Academy of Sciences. 82 (2): 488-92. doi:10.1073/pnas.82.2.488. PMC 397064 $2. PMID 3881765.
- Wells, J. A.; Estell, D. A. (1988). "Subtilisin--an enzyme designed to be engineered". Trends in Biochemical Sciences. 13 (8): 291-297. doi:10.1016/0968-0004(88)90121-1. PMID 3154281.
- Karnik, Abhijit; Karnik, Rucha; Grefen, Christopher (2013). "SDM-Assist software to design site-directed mutagenesis primers introducing "silent" restriction sites". BMC Bioinformatics. 14 (1): 105. doi:10.1186/1471-2105-14-105. ISSN 1471-2105. PMC 3644487 $2. PMID 23522286.
- Papworth, C., Bauer, J. C., Braman, J. and Wright, D. A. (1996). "Site-directed mutagenesis in one day with >80% efficiency". Strategies. 9 (3): 3-4.
- Edelheit, O; Hanukoglu, A; Hanukoglu, I (2009). "Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies". BMC Biotechnol. 9: 61. doi:10.1186/1472-6750-9-61. PMC 2711942 $2. PMID 19566935.
- Cerchione, Derek; Loveluck, Katherine; Tillotson, Eric L.; Harbinski, Fred; DaSilva, Jen; Kelley, Chase P.; Keston-Smith, Elise; Fernandez, Cecilia A.; Myer, Vic E.; Jayaram, Hariharan; Steinberg, Barrett E. (16 Nisan 2020). [10.1371/journal.pone.0231716 "SMOOT libraries and phage-induced directed evolution of Cas9 to engineer reduced off-target activity"]
|url=değerini kontrol edin (yardım). PLOS ONE (İngilizce). 15 (4): e0231716. doi:10.1371/journal.pone.0231716. ISSN 1932-6203. - Storici F.; Resnick MA. (2006). The delitto perfetto approach to in vivo site-directed mutagenesis and chromosome rearrangements with synthetic oligonucleotides in yeast. Methods in Enzymology. 409. ss. 329-45. doi:10.1016/S0076-6879(05)09019-1. ISBN 9780121828141. PMID 16793410.
- Storici F.; Resnick MA (2003). "Delitto perfetto targeted mutagenesis in yeast with oligonucleotides". Genetic Engineering. 25: 189-207. PMID 15260239.
- Damien Biot-Pelletier and Vincent J. J. Martin (2016). "Seamless site-directed mutagenesis of the Saccharomyces cerevisiae genome using CRISPR-Cas9". Journal of Biological Engineering. 10: 6. doi:10.1186/s13036-016-0028-1. PMC 4850645 $2. PMID 27134651.
- Xu S (20 Ağustos 2015). "The application of CRISPR-Cas9 genome editing in Caenorhabditis elegans". J Genet Genomics. 42 (8): 413-21. doi:10.1016/j.jgg.2015.06.005. PMC 4560834 $2. PMID 26336798.
- Kovács KA, Steinmann M, Halfon O, Magistretti PJ, Cardinaux JR (Kasım 2015). "Complex regulation of CREB-binding protein by homeodomain-interacting protein kinase 2" (PDF). Cellular Signalling. 27 (11): 2252-60. doi:10.1016/j.cellsig.2015.08.001. PMID 26247811.
- Reetz, M. T.; Carballeira J. D. (2007). "Iterative saturation mutagenesis (ISM) for rapid directed evolution of functional enzymes". Nature Protocols. 2 (4): 891-903. doi:10.1038/nprot.2007.72. PMID 17446890.
- Stauffer CE, Etson D (10 Ekim 1969). "The effect on subtilisin activity of oxidizing a methionine residue". Journal of Biological Chemistry. 244 (19): 5333-8. PMID 5344139.
- Estell DA, Graycar TP, Wells JA (10 Haziran 1985). "Engineering an enzyme by site-directed mutagenesis to be resistant to chemical oxidation". Journal of Biological Chemistry. 260 (11): 6518-21. PMID 3922976.
- Yury E. Khudyakov, Howard A. Fields, (Ed.) (25 Eylül 2002). Artificial DNA: Methods and Applications. CRC Press. s. 13. ISBN 9781420040166.